2 Princípios
2.1 O tidyverse
O tidyverse é um pacote do R, cuja única função é carregar outros pacotes do R. O conjunto desses pacotes forma o tidyverse. É considerado um “universo” a parte do R pois todas suas ferramentas possuem formas de uso consistentes e funcionam muito bem em conjunto.
Os princípios do tidyverse seguem abaixo.
- Eficiência algorítmica vs eficiência de trabalho. Suposição: o tempo que o estatísco gasta pensando em como realizar uma operação é mais importante do que o tempo que o computador gasta para realizar um cálculo.
- Tidy data. Princípio para arrumação de base de dados que resolve 90% dos problemas reais. O objetivo em arrumação de dados é extrair e transformar uma base de dados até que ela esteja em formato tidy. Essa é uma boa prática de análise de dados que economiza muito tempo em qualquer trabalho. Uma base de dados é considerada “tidy” se
- Cada observação é uma linha do bd.
- Cada variável é uma coluna do bd.
- Cada dado está numa célula do bd.
Utilização do operador
%>%(pipe).“No matter how complex and polished the individual operations are, it is often the quality of the glue that most directly determines the power of the system.”
– Hal Abelson- Aparato mnemônico. Pacotes baseados em teoria e API consistentes.
- Minimalidade e funções puras. Funções sem side-effects. Interagem com o mundo através de inputs e outputs. Encaixa perfeitamente com o princípio do pipe
workflow para ciência de dados
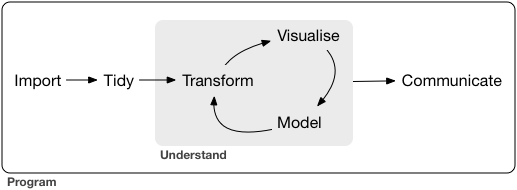
2.2 RStudio
O RStudio é a melhor IDE para usar o R. O programa possui diversas vantagens e praticamente nenhuma desvantagem. Caso tenha interesse em se ambientar e entender as características do RStudio, veja essa página.
Uma importante funcionalidade do RStudio é a possibilidade de criar projetos. Uma estrutura recomendada para organização de pacotes segue abaixo.
project/
- README.Rmd # Descrição do pacote
- set-up.R # Pacotes etc
- R/ # Código R, organizado com 0-load.R, 1-tidy.R, 2-vis.R, ...
- data/ # Dados (estruturados ou não)
- figures/ # gráficos (pode ficar dentro de output/)
- output/ # Relatórios em .Rmd, .tex etc
- project.RprojOutra possível forma de estruturar um projeto é organizando-o como um pacote do R:
project/
- README.md # Descrição do pacote
- DESCRIPTION # Metadados estruturados do pacote e dependências
- NAMESPACE # importações e exportações do pacote
- vignettes/ # Relatórios em .Rmd
- R/ # Funções do R
- data/ # Dados estruturados (tidy data)
- data-raw/ # Dados não estruturados e arqs 0-load.R, 1-tidy.R, 2-vis.R, ...
- project.RprojPara detalhes de como criar pacotes no R de forma eficiente, leia o r-pkgs. Recomendo a adoção de um critério consistente para organização de projetos. O estatístico não pode perder tempo com a estruturação das pastas, então é melhor forçar uma estrutura pré-fixada do que planejar a melhor forma de organização para cada projeto.
2.3 RMarkdown
O RMarkdown é um tipo de documento especial que contém tanto textos (em markdown) quanto códigos em R (em chunks). O markdown nada mais é do que um documento de texto com alguns padrões básicos de formatação, como negrito, itálico, títulos, subtítulos, itemização e referências cruzadas. Já os chunks são pedaços de códigos em R encapsulados por três crases “```”. Os códigos são executados sempre que o documento é processado para algum formato específico.
A utilização do RMarkdown para produção de relatórios é essencial para o estatístico pragmático. O RMarkdown possui diversas vantagens:
- Simplicidade e foco. Obriga o usuário a focar na análise e não na formatação do documento.
- Versátil. Pode ser utilizado para gerar documentos em LaTeX, Word, HTML e apresentações em beamer, pptx e HTML (de vários tipos). Pode ainda gerar sites, livros, dissertações de mestrado e até mesmo dashboards interativos.
- Reprodutível. O RMarkdown nada mais é que um arquivo de texto. Além disso, ele tenta te obrigar a fazer o documento mais autocontido possível. Assim, um documento .Rmd é fácil de compartilhar e de ser utilizado pelo receptor. Lembre-se, o receptor pode ser o futuro você! Vale enfatizar que a reprodutibilidade é considerada como um dos princípios fundamentais para a ciência. Então só de usar RMarkdown, você já está colaborando com a ciência :)
- Eficiente. É possível configurar e criar templates de análises para quaisquer tipos de aplicações e clientes.
Para detalhes sobre como utilizar o RMarkdown, leia aqui e aqui.
2.4 GitHub
O GitHub é uma plataforma online para compartilhar códigos. Projetos do GitHub são baseados no git, uma ferramenta de versionamento de software.
Utilizar o GitHub é uma boa prática de organizar projetos pois é uma forma de manter os códigos organizados e atualizados na web, sem o perigo de perder tudo acidentalmente. Esse site também é essencial para projetos colaborativos, pois aumenta a produtividade e permite que pessoas de todo lugar ajudem nos projetos. O tidyverse só é o que é hoje por conta do social coding.
Para detalhes, faça o data science toolbox.
2.5 Pipe
O operador pipe foi uma das grandes revoluções recentes do R, tornando a leitura de códigos mais lógica, fácil e compreensível. Este operador foi introduzido por Stefan Milton Bache no pacote magrittr e já existem diversos pacotes construidos para facilitar a sua utilização.
Basicamente, o operador %>% usa o resultado do seu lado esquerdo como primeiro argumento da função do lado direito. Só isso!
Para usar o operador %>%, primeiramente instale o pacote magrittr.
install.packages("magrittr")e carregá-lo com a função library()
library(magrittr)Feito isso, vamos testar o operador calculando a raiz quadrada da soma de alguns números.
x <- c(1, 2, 3, 4)
x %>% sum %>% sqrt## [1] 3.162278O caminho que o código acima seguiu foi enviar o objeto x como argumento da função sum() e, em seguida, enviar a saida da expressão sum(x) como argumento da função sqrt(). Observe que não é necessário colocar os parênteses após o nome das funções.
Se escrevermos esse cálculo na forma usual, temos o seguinte código:
sqrt(sum(x))## [1] 3.162278A princípio, a utilização do %>% não parece trazer grandes vantagens, pois a expressão sqrt(sum(x)) é facilmente compreendida. No entanto, se tivermos um grande número de funções aninhadas, a utilização do pipe transforma um código confuso e difícil de ser lido em algo simples e intuitivo. Como exemplo, imagine que você precise escrever uma receita de um bolo usando o R, e cada passo da receita é uma função:
esfrie(asse(coloque(bata(acrescente(recipiente(rep("farinha", 2), "água", "fermento", "leite", "óleo"), "farinha", até = "macio"), duração = "3min"), lugar = "forma", tipo = "grande", untada = T), duração = "50min"), "geladeira", "20min")Tente entender o que é preciso fazer. Nada fácil, correto? Agora escrevemos usando o operador %>%:
recipiente(rep("farinha", 2), "água", "fermento", "leite", "óleo") %>%
acrescente("farinha", até = "macio") %>%
bata(duração = "3min") %>%
coloque(lugar = "forma", tipo = "grande", untada = T) %>%
asse(duração = "50min") %>%
esfrie("geladeira", "20min")Agora o código realmente parece uma receita de bolo.
Para mais informações sobre o pipe e exemplos de utilização, visite a página Ceci n’est pas un pipe.